解决R语言报错:Error in y + 1:non-numeric argument to binary operator
因为花了2天半才解决,中间痛苦的寻找,记录一下解决的流程与经验
报错信息:
1Error in y + 1 : non-numeric argument to binary operator
报错原因:
数据不是可计算的 numeric 或 integer 类型
原代码:
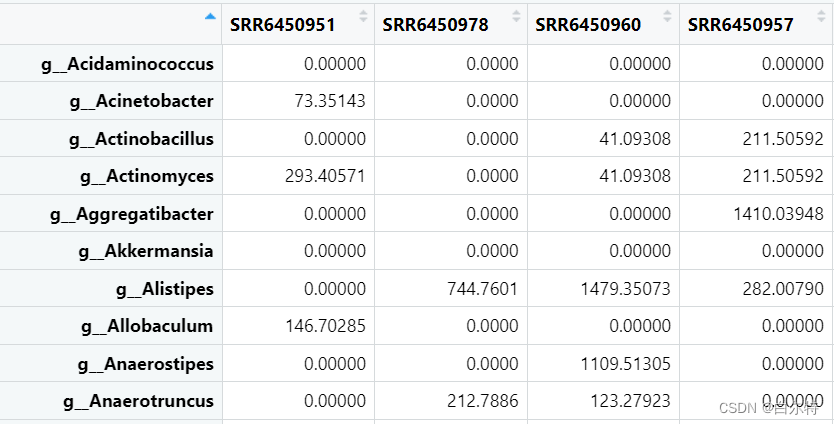
a = read.table(file = study.txt", sep = "\t", header = T, row.names = 1 ) class(a[3, 3]) # integer aa = t(d) class(aa[3, 3]) # character b = sparcc(aa) # 出现报错 Error in y + 1 : non-numeric argument to binary operator
报错原因解析:
1. 转置后数据类型变为character,因为numeric数据中存在character类型的脏数据
(原因:转置函数t() 是先将dataframe转换为矩阵matrix,而matrix只有一种数据类型。所以如果存在character,所有数据都会被转换成character)
如何发现是否有character脏数据:
read.table设置参数colClasses = “numeric”(确保数据框内只有numeric类型)
a = read.table(file = study.txt", sep = "\t", header = T, row.names = 1 colClasses = "numeric" # 添加的参数 ) # 出现报错 Error in scan(file = file, what = what, sep = sep, quote = quote, dec = dec, : scan() expected 'a real', got 'f__Cenarchaeaceae'
报错意为 数据框内存在“f__Cenarchaeaceae”,不属于numeric
查看txt内部

2. 引入character脏数据的原因
# 后续分析需要:设置data第一列列名为空格 genus <- data[1] colnames(genus) <- " " # 根据列名提取子集 a <- subset(data, select = (disID[, 1]))
subset()函数将列名为 空格blank 的也提取了,导致了character脏数据的进入
总结
到此这篇关于解决R语言报错:Error in y + 1:non-numeric argument to binary operator的文章就介绍到这了,更多相关R语言报错1Error in y + 1 内容请搜索脚本之家以前的文章或继续浏览下面的相关文章希望大家以后多多支持脚本之家!
相关文章

详解R语言中的多项式回归、局部回归、核平滑和平滑样条回归模型
这篇文章主要介绍了R语言中的多项式回归、局部回归、核平滑和平滑样条回归模型,本文给大家介绍的非常详细,对大家的学习或工作具有一定的参考借鉴价值,需要的朋友可以参考下2021-03-03
解决R语言中install_github中无法安装遇到的问题
这篇文章主要介绍了解决R语言中install_github中无法安装遇到的问题,具有很好的参考价值,希望对大家有所帮助。一起跟随小编过来看看吧2021-04-04











最新评论